Mars 2024 var jeg så heldig å få reise til Glasgow for å besøke professor Emma Thomson og hennes kolleger ved Centre for Virus Research (CVR) – et samarbeid mellom Medical Research Council og University of Glasgow. Senterets hovedfokus er å hindre spredning av virale sykdommer gjennom tverrfaglig samarbeid mellom human- og veterinærmedisin og ulike laboratoriedisipliner. Bygget de holder til i er relativt nytt, og virket å være godt tilrettelagt for dette med flere sosiale soner og lett tilgang på nødvendig ekspertise. Senteret har blant annet flere BSL3-laboratorier, celledyrkningsfasiliteter, avanserte mikroskoper (f.eks. to kryo-EM), HTS-instrumenter (både Illumina og ONT) og dyrestaller (en stall er forbeholdt mygg).

Hovedinngangen til senteret.

Etasjeoversikt på senteret.
Ettersom senteret fokuserer mye på nye virus og/eller nye virusmutasjoner lener de seg mest på metagenomikk eller «probe-based capture» i stedet for amplikonbaserte HTS-prosedyrer. De benytter i hovedsak tre ulike anrikningspaneler: Et pan-viralt, et for arbovirus, og et for respiratoriske virus. Særlig interessant var det å diskutere utfordringer rundt terskelverdier (cut-offs) for hva som skal regnes som funn og hva som ikke skal det, samt metoder for kvalitetssikring og kontaminasjonskontroll. Her har de blant annet utviklet en egen metode med «spiking» av et utvalg brønner, og en bioinformatisk pipeline for å følge med på hvilke brønner det «spikede» materialet eventuelt smitter over til, se bilde under. Da jeg spurte dem om sine beste råd for å lykkes med probebasert capture av RNA-virus var det følgende: (1) Vurdér å bruke en mosquito pipetteringsrobot (gir lavere dødvolum og høyere coverage), (2) Kjøp en UPS til varmeblokken som benyttes til hybridisering, og (3) Vurdér SMART 9N-protokollen.
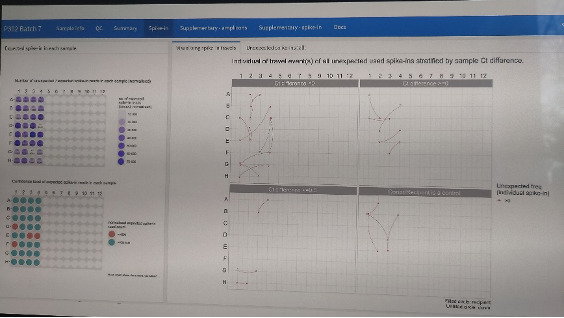
Skjermbilde fra QC-program for kontroll av kontaminering.

Mosquito pipetteringsrobot.
Andre metoder senteret benytter er «single cell sequencing» og «spacial transcriptomics». Ved hjelp av sistnevnte metode benyttes et instrument fra Nanostring for å studere infiserte celler i 3D. Se under for hvordan Dr Ana da Silva Filipe forklarte meg forskjellen på de ulike metodene som kan benyttes for å studere infiserte celler.
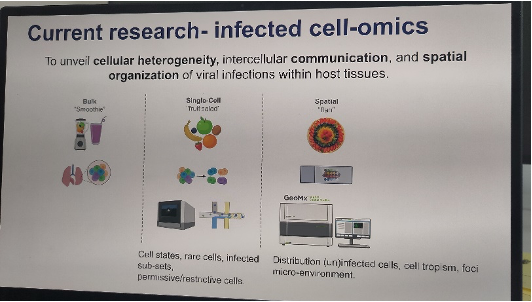
Skjermbilde fra Dr. Ana de Silva Filipes presentasjon av «spatial transcriptomics»

Danny demonstrerer Nanostring-instrumentet.
Ved siden av å få omvisning på laboratoriene og ha samtaler med et bredt utvalg av ansatte, fikk jeg også delta på tre tverrfaglige seminarer hvor jeg på et av dem fikk presentere mitt eget arbeid. Etter dette fikk jeg mange gode tilbakemeldinger og råd for videre arbeid.